Aantekeningen BTMOL - MBT
WC02
DNA wordt gesynthetiseerd van 5’ 3’
1000bp = 1kb
TA (annealing temperatuur) Laagste TM minus 5°C
Te hoge TA = primers binden niet.
Te lage TA = aspecifiek PCR product, primers binden niet alleen op de bedoelde sequentie.
TM = smelttemperatuur Aanbevolen is 5°C of minder verschil tussen de primers.
PCR programma:
- Denaturatie
- Annealing
- Elongatie / extensie
Extensiesnelheid aantal bp / minuten
Mismatch = niet-complementaire basen
WC03
GFP Green Fluorescent Protein
Vector = een DNA drager
Insert = DNA fragment
Digestie DNA knippen op specifieke plekken met restrictie-enzymen.
Ligatie DNA fragment in een plasmide bouwen.
Transformatie Het construct (vector+insert) wordt opgenomen in de bacteriën.
Selectie Antibioticum toevoegen aan de bacteriekweekplaten, hierdoor groeien alleen de
bacteriën die een plasmide (met resistentie-gen) hebben opgenomen.
Identificatie Controleren of de bacteriën het juiste construct hebben opgenomen.
Amplificatie Het opkweken van de bacteriën die het juiste construct hebben opgenomen.
Palindroom sequentie Je kunt de sequentie 180° draaien en dan staat er hetzelfde.
Voorbeeld: EcoRI
‘5 GAATTC 3’
‘3 CTTAAG 5’
Restrictiesite = herkenningssite van restrictie-enzym.
Sticky end Restrictie-enzymen knippen beide DNA-strengen op verschillende plaatsen en creëren
daarmee een ssDNA overhang.
Blunt end Restrictie-enzymen knippen het DNA op dezelfde plek waardoor het DNA overal
dubbelstrengs blijft.
,MCS (Multiple Cloning Site) Kan ook polylinker worden genoemd. Het is een sequentie binnen een
plasmide waar een aantal restrictiesites dicht op elkaar voorkomen, van restrictie-enzymen die
slechts eenmaal binnen de sequentie van deze plasmide knippen.
Kan worden gebruikt om een enzym uit te zoeken wat gebruikt kan worden om insert in vector te
ligeren.
Bij expressie-vectoren is het belangrijk dat de MCS volgt op een promotor en dat het readingframe
van de start van translatie naar het eiwitcoderende gedeelte van insert in frame ononderbroken door
een stopcodon doorloopt.
Omgedraaide oriëntatie Kan voorkomen als je de sequentie aan beide uiteindelijk met hetzelfde
restrictie-enzym knipt en dat in de plasmide wilt ligeren.
Berekening ng met insert-vector-ratio ng plasmide / bp plasmide * bp insert * ratio / conc insert
Componenten ligatie-reactie:
Vector
Insert
DNA ligase
Buffer met juiste pH en ionen en ATP
MilliQ (aanvullen)
Restrictie-endonucleases Enzymen die binnen in de sequentie nucleotiden afsplitsen.
Restrictie-exonucleases Enzymen die aan de uiteinden van het DNA nucleotiden afsplitsen.
In een primer worden meestal 2 of 3 extra nucleotiden toegevoegd aan de 5’ kant om restrictiesites
te introduceren en houvast te geven aan hun herkenningssequentie.
Codon = de code van 3 nucleotiden dat voor een specifiek aminozuur codeert.
Reading frame = van startcodon naar stopcodon van een sequentie.
In frame als van start naar stop er steeds drie bases zijn die coderen voor een aminozuur.
Is dit niet het geval, dan ligt de sequentie uit frame Er wordt een onlogische en onfunctionele
eiwitketen gevormd met een vroegtijdig einde, omdat het ribosoom bij toeval een stopcodon
tegenkomt.
Ongeknipt DNA Beweegt sneller door de gel omdat het DNA nog supercoiled is en daardoor
compacter op elkaar zit.
Geknipt DNA Beweegt langzamer door de gel omdat het DNA geknipt is. Het wordt dan relaxed
DNA en het is minder compact dan voorheen.
WC04
In vivo = in levende cellen
In vitro = in een epje
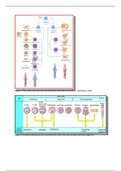
Transformeren Het genetisch veranderen van een organisme; toevoegen, weghalen of aanpassen
, van de aanwezige genetische informatie.
In bacteriën = het opnemen van een plasmide met een DNA sequentie die normaliter niet bij deze
bacterie gevonden wordt.
Chemische transformatie (heatshock) De celwand van bacteriën verzwakken door ze bij een lage
temperatuur in een buffer met bepaalde zouten te incuberen. Hierna plasmide toevoegen en
incuberen bij hoge temperatuur. DNA wordt hierdoor opgenomen in de bacteriën.
Elektroporatie Bacteriën blootstellen aan een elektrisch veld met hoge voltage waardoor het DNA
opgenomen kan worden.
Competente cellen Bacteriën die geschikt zijn (gemaakt) om vreemd DNA op te nemen.
Selectief kweekmedium Antibioticum toevoegen zodat bacteriën sterven, tenzij ze een eiwit
maken wat de bacterie resistent maakt. De bacterie moet dan een plasmide bevatten waarop een
dergelijk eiwit gecodeerd wordt, zo blijven alleen de bacteriën leven die het plasmide bevatten.
Selectiedruk = in aanwezigheid van antibioticum de bacteriekweek opkweken.
Transformatie Transformeren van prokaryote cellen
Transfectie Transformeren van eukaryote cellen
DNA is ingebracht in een eukaryote cel, hierna moet de cel eerst delen zodat het DNA in de celkern
terecht kan komen. Dan wordt het DNA tot expressie gebracht.
Gewone PCR RT-qPCR
Pas op het einde kijken of er een product is Tijdens het amplificatieproces wordt er na elke
gevormd. cyclus gemeten hoeveel DNA er aanwezig is.
Het wordt gerund op een agarosegel, gekleurd Er wordt een kleurstof toegevoegd die
met een kleurstof die het DNA bindt. fluoresceert als er DNA producten gevormd
Bijvoorbeeld: worden. Na elke extensiestap wordt een meting
SYBR Safe gedaan met fluorescentiecamera.
Ethidiumbromide
Methoden om te zorgen dat vorming dsDNA
leidt tot toename fluorescentie:
SYBR Green
Probe-based / TaqMan
Threshold Wordt in het exponetiële gebied van de curve gezet omdat er daar nog voldoende
reactiecomponenten aanwezig zijn om elke cyclus een verdubbeling van het DNA te krijgen.
Dit meetgebied = maat voor origineel aanwezige hoeveelheid input DNA.
Ct-waardes = de lijn die kruist met threshold aflezen waardoor je de hoeveelheid cycli weet.
Lagere Ct-waarde Dan krijg je een hogere expressie van je sequentie van interesse (hogere
hoeveelheid input DNA).
Rn = Normalized Reporter Value De gemeten fluorescentiewaarde minus de achtergrond.
WC02
DNA wordt gesynthetiseerd van 5’ 3’
1000bp = 1kb
TA (annealing temperatuur) Laagste TM minus 5°C
Te hoge TA = primers binden niet.
Te lage TA = aspecifiek PCR product, primers binden niet alleen op de bedoelde sequentie.
TM = smelttemperatuur Aanbevolen is 5°C of minder verschil tussen de primers.
PCR programma:
- Denaturatie
- Annealing
- Elongatie / extensie
Extensiesnelheid aantal bp / minuten
Mismatch = niet-complementaire basen
WC03
GFP Green Fluorescent Protein
Vector = een DNA drager
Insert = DNA fragment
Digestie DNA knippen op specifieke plekken met restrictie-enzymen.
Ligatie DNA fragment in een plasmide bouwen.
Transformatie Het construct (vector+insert) wordt opgenomen in de bacteriën.
Selectie Antibioticum toevoegen aan de bacteriekweekplaten, hierdoor groeien alleen de
bacteriën die een plasmide (met resistentie-gen) hebben opgenomen.
Identificatie Controleren of de bacteriën het juiste construct hebben opgenomen.
Amplificatie Het opkweken van de bacteriën die het juiste construct hebben opgenomen.
Palindroom sequentie Je kunt de sequentie 180° draaien en dan staat er hetzelfde.
Voorbeeld: EcoRI
‘5 GAATTC 3’
‘3 CTTAAG 5’
Restrictiesite = herkenningssite van restrictie-enzym.
Sticky end Restrictie-enzymen knippen beide DNA-strengen op verschillende plaatsen en creëren
daarmee een ssDNA overhang.
Blunt end Restrictie-enzymen knippen het DNA op dezelfde plek waardoor het DNA overal
dubbelstrengs blijft.
,MCS (Multiple Cloning Site) Kan ook polylinker worden genoemd. Het is een sequentie binnen een
plasmide waar een aantal restrictiesites dicht op elkaar voorkomen, van restrictie-enzymen die
slechts eenmaal binnen de sequentie van deze plasmide knippen.
Kan worden gebruikt om een enzym uit te zoeken wat gebruikt kan worden om insert in vector te
ligeren.
Bij expressie-vectoren is het belangrijk dat de MCS volgt op een promotor en dat het readingframe
van de start van translatie naar het eiwitcoderende gedeelte van insert in frame ononderbroken door
een stopcodon doorloopt.
Omgedraaide oriëntatie Kan voorkomen als je de sequentie aan beide uiteindelijk met hetzelfde
restrictie-enzym knipt en dat in de plasmide wilt ligeren.
Berekening ng met insert-vector-ratio ng plasmide / bp plasmide * bp insert * ratio / conc insert
Componenten ligatie-reactie:
Vector
Insert
DNA ligase
Buffer met juiste pH en ionen en ATP
MilliQ (aanvullen)
Restrictie-endonucleases Enzymen die binnen in de sequentie nucleotiden afsplitsen.
Restrictie-exonucleases Enzymen die aan de uiteinden van het DNA nucleotiden afsplitsen.
In een primer worden meestal 2 of 3 extra nucleotiden toegevoegd aan de 5’ kant om restrictiesites
te introduceren en houvast te geven aan hun herkenningssequentie.
Codon = de code van 3 nucleotiden dat voor een specifiek aminozuur codeert.
Reading frame = van startcodon naar stopcodon van een sequentie.
In frame als van start naar stop er steeds drie bases zijn die coderen voor een aminozuur.
Is dit niet het geval, dan ligt de sequentie uit frame Er wordt een onlogische en onfunctionele
eiwitketen gevormd met een vroegtijdig einde, omdat het ribosoom bij toeval een stopcodon
tegenkomt.
Ongeknipt DNA Beweegt sneller door de gel omdat het DNA nog supercoiled is en daardoor
compacter op elkaar zit.
Geknipt DNA Beweegt langzamer door de gel omdat het DNA geknipt is. Het wordt dan relaxed
DNA en het is minder compact dan voorheen.
WC04
In vivo = in levende cellen
In vitro = in een epje
Transformeren Het genetisch veranderen van een organisme; toevoegen, weghalen of aanpassen
, van de aanwezige genetische informatie.
In bacteriën = het opnemen van een plasmide met een DNA sequentie die normaliter niet bij deze
bacterie gevonden wordt.
Chemische transformatie (heatshock) De celwand van bacteriën verzwakken door ze bij een lage
temperatuur in een buffer met bepaalde zouten te incuberen. Hierna plasmide toevoegen en
incuberen bij hoge temperatuur. DNA wordt hierdoor opgenomen in de bacteriën.
Elektroporatie Bacteriën blootstellen aan een elektrisch veld met hoge voltage waardoor het DNA
opgenomen kan worden.
Competente cellen Bacteriën die geschikt zijn (gemaakt) om vreemd DNA op te nemen.
Selectief kweekmedium Antibioticum toevoegen zodat bacteriën sterven, tenzij ze een eiwit
maken wat de bacterie resistent maakt. De bacterie moet dan een plasmide bevatten waarop een
dergelijk eiwit gecodeerd wordt, zo blijven alleen de bacteriën leven die het plasmide bevatten.
Selectiedruk = in aanwezigheid van antibioticum de bacteriekweek opkweken.
Transformatie Transformeren van prokaryote cellen
Transfectie Transformeren van eukaryote cellen
DNA is ingebracht in een eukaryote cel, hierna moet de cel eerst delen zodat het DNA in de celkern
terecht kan komen. Dan wordt het DNA tot expressie gebracht.
Gewone PCR RT-qPCR
Pas op het einde kijken of er een product is Tijdens het amplificatieproces wordt er na elke
gevormd. cyclus gemeten hoeveel DNA er aanwezig is.
Het wordt gerund op een agarosegel, gekleurd Er wordt een kleurstof toegevoegd die
met een kleurstof die het DNA bindt. fluoresceert als er DNA producten gevormd
Bijvoorbeeld: worden. Na elke extensiestap wordt een meting
SYBR Safe gedaan met fluorescentiecamera.
Ethidiumbromide
Methoden om te zorgen dat vorming dsDNA
leidt tot toename fluorescentie:
SYBR Green
Probe-based / TaqMan
Threshold Wordt in het exponetiële gebied van de curve gezet omdat er daar nog voldoende
reactiecomponenten aanwezig zijn om elke cyclus een verdubbeling van het DNA te krijgen.
Dit meetgebied = maat voor origineel aanwezige hoeveelheid input DNA.
Ct-waardes = de lijn die kruist met threshold aflezen waardoor je de hoeveelheid cycli weet.
Lagere Ct-waarde Dan krijg je een hogere expressie van je sequentie van interesse (hogere
hoeveelheid input DNA).
Rn = Normalized Reporter Value De gemeten fluorescentiewaarde minus de achtergrond.