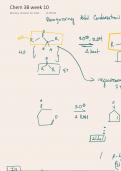
~ in Eukaryote
Molekulare Protein biosynthese
Genetik
Struktur der DNA
↳ Prozess in Zellen ,
bei dem anhand
Information (DNA) Proteine aus
DNA (Desoxyribonukleinsäurel enthält Geninformationen
,
·
gebildet werden >
-
Genexpressio
↳ besteht aus Verbundenen Bausteinen -3
Nukleotiden
·
in
jeder Zelle (in Form von Chromosomen t im Zellkern) -Problem :
·
Doppelhelix aufgebaut DNA-Replikation -
DNA bei Erkaryoten) im Zellh
↳ identische Verdopplung -Proteinbiosynthese an Ribosome
der DNA bei Zellteilung -
DNA aus Nekleotide Protein
Nukleotid Zusammensetzung ·
1 DNA Doppelhelix wird ↳ Prozess der Proteinbiosynthes
durch Enzym Helicase
strukturelle Funktion 1
auf
↳ 1 Transkription :
Zucker DNA-Sequenz
-
an
.
:
Informationen d
genetische
gespaltenentwend
eine
·
Desoxyribose (besteht Kohlenstoff-
>
aus
-
·
die für Synthese eines Prote
fünf Kohlenstoffatomen) atome
CReplikationsursprung RNA
benötigt werden in
durch nummeriert
↳ ohne Sauerstoff" umgeschrieben
von 1 bis 5 ·
2 Helicase trennt beide
2 Kohlen- (mRNA)
messenger RNA
·
>
-
fehlt am als
Centwindet
.
Einzelstränge Voneinander auf
dem Zellkern in das
Stoffatom >
-
entsteht
Replikationsgabel
aus
·
in Replikationsgabel angelagerte Proteine verhindern , Cytoplasma transportiert
das sich wieder verbinden
Einzelstränge
Phosphatgruppe :
Strukturelle Funktion
·
Jedes zuckermolekül .
3 Synthese neuer
DNA-Stränge durch Enzym
mit einem
DNA Polymerase
je
L verlängert DNA-Stränge in dem es einzelne
Phosphatrest verbunden
Nukleotide an diese bindet
(Verhältnis 1 : 1) Transkription : Übertragung Base
4
. Enzym Primase stellt Primer her (Startermoleküll ↳ in Zellkern der Gene in MRN
Primer Nucleotidsequenz
Komplementär
ChichtgesamteDN
↳ zu kurzer
Basen : tragen genetische Information der alten Einzelstränge gebildet ① Initiation :
>
-
gen
↳ an 31-Ende des primers DNA-Polymerase
bindet
Gens (DNA-Ab
Am
Anfang des
·
Basen Komplementär Nucleotidsequenz des
jeweiligen
stickstoffhaltige
zur
·
vier
Nucleotide
Kurze DNA-Basensequenz-Pr
alten Einzelstranges freie (aus Cellplasmal
↳ Adenin (A) 1 Kohlenstoff-
RNA-Polymerase
↳ bindet an >
(T)
·
↳ entstent neuer DNA strang
-
Stickstoff-Ring
↳
Thymin bewest sich entlang DNA-D
·
.
5 wird
↳ Guanin (6) DNA-Polymerase kann neuen
Einzelstrang nur in ·
innerhalb Polymerase
(d
( 5-3' Richtung Synthetisieren entwunden und in Einzelst
↳
Cytosin ↳ 2 Kohlenstoff-
Leitstrang : Kontinuierliche Verlängerung
bindt Stickstoff-Ringe
↳
aufgetrennt (Wasserstoffbrüch
·
eine Base an Zucker ↳ Folgestrang: diskontinuierliche Verlängerung ↳
blasenartige Öffnung Dopp
davor + dahinter bleibt D
↳ Diskontinuierliche Synthese :
Doppelstränge verlaufen antip
>
-
Nukleotid Aufbau Einzelstrang wird in entgegengesetzte Richtung
(3'-5' Ausrichtung
-
Replikationsgabel weg) synthetisiert > Folgestrang
,
RNA-Poly
Desoxyribose , Phosphatgruppe und Base Loon -
↳ zusammen
liest von 3'-5' Richtung
neue Primer gebildet
-
=> Nukleotid 3-Enden
-
an hängt DNA-Polymerase Nucleotide an
-
Base an 1-Kohlenstoff-Atom des Zuckers gebunden zwischen Primern Komplementär
Elongation
entstehen zum alten
2
-
:
-
Phosphatgruppe an S-Kohlenstoff-Atom Einzelstrang DNA-Stücke neuer Strang
RNA-Polymerase 3'-5-Richt
·
↳
Lücken -
Okazaki-Fragmente
-
DNA besteht aus Kette vieler Nucleotide
codogener Strang abgelesen
=>
Polynucleotid 6 .
Primer durch Enzym abgebaut
·
RNA-Nukleotice Lagern sich
Lücken komplementäre Nucleotide in 5-3-Richtung Ende
durch
gefüllt
·
an
okazahiFragmentedurchmisasans
y'-Ende
·
Wachsenden dran
·
Nucleotide über Phosphatgruppe miteinander verbunden
·
durch RNA-Polymerase zu
5-Kohlenstoffatom
Phosphatgruppe eines Nucleotids
·
am
mit 3-Kohlenstoff-Atom des
folgenden Nucleotids verbunden 7
7 Zwei identische DNA-Doppelstränge MRNA-Strang verbunden
Mechanismus : 3 Termination :
Semivonservativer
>
Am 3-Ende freie Ol-Gruppe
>
-
Nach Ablesen
-
reißverschlossatig
·
in zwei
DNA-Doppelstrang teilt sich
-
freie RNA-Polymerase erreicht
> Am 5-Ende
Phosphatgruppe Längshälften (die mit Nutleotiden zwei DNA-Doppelsträngen
·
-
zu
ergänzt werden (bestimmte Basensequenz S
↳ in neu gebildeten DNA-Strängen bleibt jeweils Hälfte ·
läst sich von DNA -MRNA
Raumstruktur DNA (semi) der Ausgangs-DNA (honserviert)
freigesetzt
erhalten
wird
DNA-Bereich schließt sich
·
DNA/RNA
-Modell von Watson & Crick ·
mRNA-Strang verlässt Z
Einzelstrang Doppelstrang
↳ DNA Aufbau gleicht einer Leiter RNA :
codiertende Funktion
Nukleotiden)
-
(zwei Ketten mit LmRNA Transport einer Kopie)
Molekulare Protein biosynthese
Genetik
Struktur der DNA
↳ Prozess in Zellen ,
bei dem anhand
Information (DNA) Proteine aus
DNA (Desoxyribonukleinsäurel enthält Geninformationen
,
·
gebildet werden >
-
Genexpressio
↳ besteht aus Verbundenen Bausteinen -3
Nukleotiden
·
in
jeder Zelle (in Form von Chromosomen t im Zellkern) -Problem :
·
Doppelhelix aufgebaut DNA-Replikation -
DNA bei Erkaryoten) im Zellh
↳ identische Verdopplung -Proteinbiosynthese an Ribosome
der DNA bei Zellteilung -
DNA aus Nekleotide Protein
Nukleotid Zusammensetzung ·
1 DNA Doppelhelix wird ↳ Prozess der Proteinbiosynthes
durch Enzym Helicase
strukturelle Funktion 1
auf
↳ 1 Transkription :
Zucker DNA-Sequenz
-
an
.
:
Informationen d
genetische
gespaltenentwend
eine
·
Desoxyribose (besteht Kohlenstoff-
>
aus
-
·
die für Synthese eines Prote
fünf Kohlenstoffatomen) atome
CReplikationsursprung RNA
benötigt werden in
durch nummeriert
↳ ohne Sauerstoff" umgeschrieben
von 1 bis 5 ·
2 Helicase trennt beide
2 Kohlen- (mRNA)
messenger RNA
·
>
-
fehlt am als
Centwindet
.
Einzelstränge Voneinander auf
dem Zellkern in das
Stoffatom >
-
entsteht
Replikationsgabel
aus
·
in Replikationsgabel angelagerte Proteine verhindern , Cytoplasma transportiert
das sich wieder verbinden
Einzelstränge
Phosphatgruppe :
Strukturelle Funktion
·
Jedes zuckermolekül .
3 Synthese neuer
DNA-Stränge durch Enzym
mit einem
DNA Polymerase
je
L verlängert DNA-Stränge in dem es einzelne
Phosphatrest verbunden
Nukleotide an diese bindet
(Verhältnis 1 : 1) Transkription : Übertragung Base
4
. Enzym Primase stellt Primer her (Startermoleküll ↳ in Zellkern der Gene in MRN
Primer Nucleotidsequenz
Komplementär
ChichtgesamteDN
↳ zu kurzer
Basen : tragen genetische Information der alten Einzelstränge gebildet ① Initiation :
>
-
gen
↳ an 31-Ende des primers DNA-Polymerase
bindet
Gens (DNA-Ab
Am
Anfang des
·
Basen Komplementär Nucleotidsequenz des
jeweiligen
stickstoffhaltige
zur
·
vier
Nucleotide
Kurze DNA-Basensequenz-Pr
alten Einzelstranges freie (aus Cellplasmal
↳ Adenin (A) 1 Kohlenstoff-
RNA-Polymerase
↳ bindet an >
(T)
·
↳ entstent neuer DNA strang
-
Stickstoff-Ring
↳
Thymin bewest sich entlang DNA-D
·
.
5 wird
↳ Guanin (6) DNA-Polymerase kann neuen
Einzelstrang nur in ·
innerhalb Polymerase
(d
( 5-3' Richtung Synthetisieren entwunden und in Einzelst
↳
Cytosin ↳ 2 Kohlenstoff-
Leitstrang : Kontinuierliche Verlängerung
bindt Stickstoff-Ringe
↳
aufgetrennt (Wasserstoffbrüch
·
eine Base an Zucker ↳ Folgestrang: diskontinuierliche Verlängerung ↳
blasenartige Öffnung Dopp
davor + dahinter bleibt D
↳ Diskontinuierliche Synthese :
Doppelstränge verlaufen antip
>
-
Nukleotid Aufbau Einzelstrang wird in entgegengesetzte Richtung
(3'-5' Ausrichtung
-
Replikationsgabel weg) synthetisiert > Folgestrang
,
RNA-Poly
Desoxyribose , Phosphatgruppe und Base Loon -
↳ zusammen
liest von 3'-5' Richtung
neue Primer gebildet
-
=> Nukleotid 3-Enden
-
an hängt DNA-Polymerase Nucleotide an
-
Base an 1-Kohlenstoff-Atom des Zuckers gebunden zwischen Primern Komplementär
Elongation
entstehen zum alten
2
-
:
-
Phosphatgruppe an S-Kohlenstoff-Atom Einzelstrang DNA-Stücke neuer Strang
RNA-Polymerase 3'-5-Richt
·
↳
Lücken -
Okazaki-Fragmente
-
DNA besteht aus Kette vieler Nucleotide
codogener Strang abgelesen
=>
Polynucleotid 6 .
Primer durch Enzym abgebaut
·
RNA-Nukleotice Lagern sich
Lücken komplementäre Nucleotide in 5-3-Richtung Ende
durch
gefüllt
·
an
okazahiFragmentedurchmisasans
y'-Ende
·
Wachsenden dran
·
Nucleotide über Phosphatgruppe miteinander verbunden
·
durch RNA-Polymerase zu
5-Kohlenstoffatom
Phosphatgruppe eines Nucleotids
·
am
mit 3-Kohlenstoff-Atom des
folgenden Nucleotids verbunden 7
7 Zwei identische DNA-Doppelstränge MRNA-Strang verbunden
Mechanismus : 3 Termination :
Semivonservativer
>
Am 3-Ende freie Ol-Gruppe
>
-
Nach Ablesen
-
reißverschlossatig
·
in zwei
DNA-Doppelstrang teilt sich
-
freie RNA-Polymerase erreicht
> Am 5-Ende
Phosphatgruppe Längshälften (die mit Nutleotiden zwei DNA-Doppelsträngen
·
-
zu
ergänzt werden (bestimmte Basensequenz S
↳ in neu gebildeten DNA-Strängen bleibt jeweils Hälfte ·
läst sich von DNA -MRNA
Raumstruktur DNA (semi) der Ausgangs-DNA (honserviert)
freigesetzt
erhalten
wird
DNA-Bereich schließt sich
·
DNA/RNA
-Modell von Watson & Crick ·
mRNA-Strang verlässt Z
Einzelstrang Doppelstrang
↳ DNA Aufbau gleicht einer Leiter RNA :
codiertende Funktion
Nukleotiden)
-
(zwei Ketten mit LmRNA Transport einer Kopie)